Les grandes découvertes de la génétique (en lecture)
La génétique est une discipline relativement jeune, puisque les premières lois de l'hérédité n'ont été découvertes qu'il y a 140 ans. Encore ces bases fondamentales n'ont-elles été jetées que grâce à la perspicacité d'un scientifique très en avance sur son temps, Grégor Mendel. Les lois qu'il décrivit furent d'ailleurs ignorées de la plupart de ses contemporains, oubliées durant plus de 40 ans, avant d'être redécouvertes au début de ce siècle, leur importance enfin reconnue et leurs principes enrichis. Mais ce n'est que grâce à la découverte de la structure de l'ADN dans les années 1950-1960, puis à l'invention du formidable outil que constitue le génie génétique vers 1975, que cette discipline a pu réaliser les fantastiques progrès qui ont bouleversé tout le champ de la biologie et de la médecine.
La génétique mendélienne
La génétique morganienne
La génétique moléculaire
La révolution du génie génétique
La génétique mendélienne
Les premières bases de la génétique ont été établies vers 1860 par le moine morave Grégor Mendel, qui a observé la transmission, d'une génération à l'autre, de certains caractères apparents chez le pois. Il en a déduit des lois simples, les lois de Mendel, toujours valables aujourd'hui. Il a également émis l'hypothèse suivante : les caractères apparents transmis entre générations sont déterminés par des facteurs héréditaires qui obéissent toujours aux mêmes lois.
Cette génétique mendélienne a conduit à définir, lorsqu'on redécouvrit les lois de Mendel au début du XXe siècle, un premier vocabulaire de base :
- phénotype : ensemble des caractères apparents d'un individu,
- gènes : facteurs déterminant les caractères apparents,
- génotype : ensemble des gènes d'un individu,
- allèle : une des formes possibles d'un même gène,
- homozygote : un individu est homozygote pour un gène quand il possède deux allèles identiques de ce gène,
- hétérozygote : un individu est hétérozygote pour un gène quand il possède deux allèles différents de ce gène,
- allèle dominant ou récessif : soit un individu hétérozygote pour un gène comportant les deux allèles G et g ; si cet individu n'exprime que le caractère correspondant à l'allèle G, celui-ci sera dit "dominant" par rapport à l'allèle g "récessif".
La génétique morganienne
De 1909 à 1915, l'américain Thomas Morgan a déterminé les premiers fondements moléculaires de la génétique en étudiant la transmission héréditaire de différents caractères chez la drosophile ou mouche du vinaigre.
De ces recherches est née la théorie chromosomique de l'hérédité, dont la principale affirmation est la suivante : les gènes sont des éléments matériels portés par les chromosomes.
Ces derniers apparaissent, dans la cellule, sous forme de bâtonnets qui, au cours de la division cellulaire, subissent des processus complexes de dédoublement et de séparation.
Toutes les cellules d'une même espèce comportent 2n chromosomes (n = 23 chez l'homme), à l'exception des cellules sexuelles matures, les gamètes (spermatozoïdes et ovules), qui ne renferment que n chromosomes.
Quand un spermatozoïde à n chromosomes (paternels) féconde un ovule à n chromosomes (maternels), il en résulte un oeuf fécondé, ou zygote, à 2n chromosomes appariés (chromosomes "homologues").
Toutes les cellules issues de ce zygote par un processus de division nommé mitose, comportent n chromosomes paternels et n chromosomes maternels.
Seules les cellules précurseurs des cellules sexuelles subissent une division spéciale nommée méiose, qui aboutit à des gamètes à n chromosomes, après la séparation des chromosomes homologues.
Au cours de la méiose, a lieu un phénomène qui augmente considérablement la diversité génétique : le crossing-over (ou enjambement chromosomique), au cours duquel les chromosomes homologues s'associent puis échangent certains segments avant de se séparer. Certains gènes sont ainsi transférés d'un chromosome homologue sur l'autre. En conséquence, chacun des gamètes issus d'une même cellule précurseur possède une combinaison de gènes différente de celle des autres gamètes. Grâce au brassage génétique créé par la méiose et la fécondation, tout zygote possède un patrimoine unique de gènes paternels et maternels. De ce patrimoine résulte le phénotype.
Chaque gène occupe une position déterminée sur un chromosome. Deux gènes situés sur un même chromosome ont d'autant plus de chances de ne pas être séparés lors de la méiose - donc d'être transmis ensemble à la descendance - qu'ils sont plus proches l'un de l'autre. C'est grâce à ce principe que Morgan put établir sa théorie de l'hérédité, en comparant la transmission de plusieurs caractères chez la drosophile.
La génétique de Morgan complétait parfaitement celle de Mendel en y introduisant de nouveaux concepts. Un vocabulaire nouveau fut créé :
- distance génétique : distance entre deux gènes sur le chromosome,
- locus génique : position d'un gène sur le chromosome,
- mutation : modification du matériel héréditaire.
Restait à déterminer la nature chimique des gènes et des chromosomes. Ce fut chose faite avec l'avènement de la génétique moléculaire, qui entraîna un fulgurant essor dans la connaissance du vivant et des molécules qui le structurent.
La génétique moléculaire
Une des plus grandes découvertes du XXe siècle a été l'élucidation, au cours des années 1950, de la nature chimique du matériel génétique. Le support des gènes est constitué par la gigantesque molécule d'acide désoxyribonucléique, ou ADN, dont la structure en double hélice a été déterminée par James Watson et Francis Crick en 1961, ce qui leur valut le prix Nobel. Cette découverte a révolutionné l'étude du vivant, livrant aux biologistes une clé fondamentale pour comprendre la grammaire et le langage des cellules.
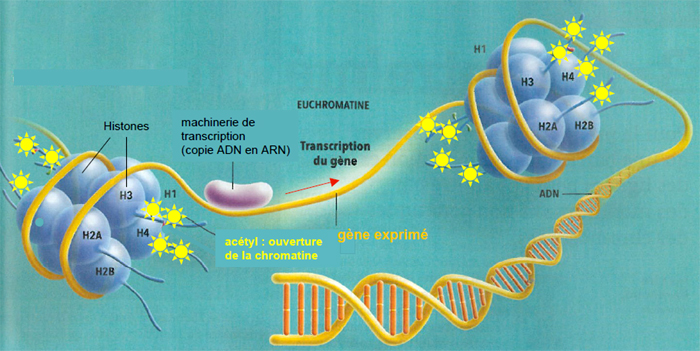
En effet, c'est précisément dans la molécule d'ADN que sont codées les instructions qui gouvernent le fonctionnement des cellules. On nomme génome l'ensemble des instructions inscrites dans le matériel génétique (généralement de l'ADN) d'un organisme. Cette molécule d'ADN forme une pelote microscopique enroulée dans le noyau des cellules (chez les organismes eucaryotes, c'est-à-dire constitués de cellules munies d'un noyau) ou dans le cytoplasme (chez les procaryotes, cellules dénuées de noyau, comme les bactéries). Une fois déroulée, la molécule d'ADN s'étire en un fil gigantesque, constitué par un enchaînement précis de caractères dans un alphabet à quatre lettres chimiques : les "bases" ou "nucléotides", symbolisés par les lettres A,T, G et C. Ces caractères forment des mots, les gènes, qui s'enchaînent le long de l'ADN.
Une des singularités de l'ADN est d'être formé de deux brins "complémentaires", semblables à une photographie et son négatif. C'est la clé du mécanisme de son dédoublement, ou réplication, qui se produit lors de la division cellulaire.
Les deux brins complémentaires se séparent alors, et le négatif de chaque brin est synthétisé. Ce processus engendre deux molécules d'ADN strictement conformes (sauf erreurs de copie ou remaniements chromosomiques) à la molécule initiale. Chacune de ces molécule-filles se distribue ensuite respectivement dans une des deux cellules issues de cette division.
Comment le message génétique codé dans cet ADN est-il lu par la cellule? Un messager intermédiaire est ici nécessaire : il s'agit de la molécule d'ARN (acide ribonucléique), qui permet le transfert de l'information génétique portée par un gène, dans le noyau cellulaire, jusqu'au cytoplasme, où cette information est traduite sous forme de molécules particulières, les protéines.
Celles-ci sont les véritables "chevilles ouvrières" de la cellule. Chaque gène code une instruction déterminant la synthèse d'une protéine particulière, chargée dans la cellule d'une tâche spécifique (on dit alors qu'un gène "code" pour une protéine donnée).
La molécule d'ARN possède une structure chimique proche de celle de l'ADN, à la différence près qu'elle ne comporte qu'un seul brin et qu'elle est constituée par un enchaînement de caractères légèrement différents, également dans un alphabet à quatre lettres : A, U, G et C. Cet ARN est synthétisé, au cours du processus de transcription, à partir d'un segment de la molécule d'ADN qui lui sert de "moule".
La molécule d'ARN alors produite est une "photocopie" d'un gène porté par l'ADN.
Cet ARN, dit "messager", migre dans le cytoplasme de la cellule. Là, il est converti en protéine au cours du processus de traduction. Une protéine est formée par un enchaînement d'autres unités chimiques : les acides aminés.
On nomme code génétique l'ensemble des règles qui font correspondre, à trois bases portées par l'ARN messager, un acide aminé déterminé de la protéine.
La révolution du génie génétique
Élucider la grammaire du vivant était certes fondamental. Encore fallait-il pouvoir aussi étudier la fonction d'un gène donné.
Jusqu'au milieu des années 1970, ce défi restait insurmontable : on ne savait pas isoler un gène déterminé, au sein de l'immense molécule d'ADN. Ce verrou fut levé grâce à l'invention, vers 1975, du génie génétique.
On désigne ainsi l'ensemble des outils qui permettent de bricoler "sur mesure" la molécule d'ADN, dans le but de purifier des gènes, de les manipuler à volonté dans le génome de différentes espèces, et de déterminer leur séquence (c'est-à-dire l'enchaînement des lettres chimiques A, T, G et C, qui constitue le message inscrit dans les gènes).
Premier outil découvert : les "enzymes de restriction", sortes de ciseaux moléculaires qui découpent l'ADN en des sites bien précis. En exploitant judicieusement ces enzymes, on a pu isoler des segments d'ADN donnés. Puis on apprit à insérer ces segments d'ADN dans des micro-organismes capables de les reproduire à l'identique en de très nombreux exemplaires, ou "clones".
Disposant ainsi du matériel génétique à étudier en quantités suffisantes, on a pu dès lors déterminer la séquence d'un gène donné, grâce aux techniques de séquençage mises au point par Paul Berg, Walter Gilbert et Frederick Sanger (prix Nobel de Chimie en 1980).
Grâce au génie génétique, l'encyclopédie de l'ADN était désormais accessible aux généticiens. Une ère nouvelle s'ouvrait : ils s'employèrent à déchiffrer ce gigantesque ouvrage. Et le rythme de leur lecture s'est considérablement accru au fil des années, parallèlement aux progrès extraordinaires des techniques disponibles.
Un prodigieux pari : le décryptage du génome
Voici seulement moins de 20 ans, l'idée de décoder l'ensemble du message inscrit dans le génome humain semblait une utopie : la molécule d'ADN humain renferme près de 3,5 milliards de signes chimiques - de quoi remplir un grimoire de plus de 500 000 pages ! - et compte quelque 100 000 gènes. Cependant, les percées spectaculaires récemment réalisées dans les procédures expérimentales de décodage de l'information génétique ont permis de relever ce défi, et l'on espère parvenir à l'objectif final du programme "génome humain" - séquencer l'intégralité du patrimoine génétique humain - dans les premières décennies du XXIe siècle.
Ces progrès sont en grande partie dus à l'automatisation des techniques (robotique et informatique) et à l'établissement de "cartes" très précises du génome humain. Dans ce domaine, la France a joué un rôle de tout premier plan, principalement grâce aux travaux de Daniel Cohen et Jean Dausset, au CEPH (Centre d'Etude du Polymorphisme Humain), de Jean Weissenbach, au laboratoire Généthon, et de Charles Auffray, du CNRS.
Les enjeux médicaux de ce nouveau champ de connaissances sont considérables : à terme, le séquençage du génome, la découverte de nouveaux gènes et l'élucidation de leur fonction conduiront à la compréhension des mécanismes des maladies génétiques, mais aussi des cancers, des maladies cardio-vasculaires, neurodégénératives et infectieuses, et devraient permettre de concevoir de nouveaux traitements.
Les cartes du génome humain
Un gigantesque défi : le séquençage du génome
Les cartes du génome humain
Ces cartes sont d'un intérêt irremplaçable pour les généticiens : elles leur permettent de localiser et d'identifier un gène en un temps record, grâce à un ensemble de repères régulièrement positionnés le long des chromosomes. On distingue trois types de cartes du génome : les cartes génétiques, les cartes physiques et les cartes des gènes.
Les cartes génétiques
La localisation d'un gène de maladie débute généralement par une phase de cartographie génétique, fondée sur l'analyse de la transmission de la maladie au sein de familles atteintes. On observe alors que les descendants malades héritent en commun d'une région chromosomique provenant des ascendants qui transmettent la maladie, tandis que les descendants sains héritent de l'allèle "non atteint" de cette même région. On peut ainsi cerner la région chromosomique contenant le gène responsable de la maladie.
Toutefois, pour étudier la transmission des chromosomes parentaux, il faut pouvoir disposer d'une carte génétique de référence, sur laquelle ont été placés régulièrement de très nombreux repères permettant de distinguer les copies chromosomiques issues de chaque parent, voire de chaque grand-parent. Ces repères sont constitués de courtes séquences d'ADN qui présentent une très grande variabilité d'un individu à l'autre, nommées "microsatellites".
Les cartes génétiques de référence actuelles, très précises, comptent au total près de 10 000 repères, dont plus de 5 300 proviennent du laboratoire français Généthon.
Les cartes physiques
La cartographie physique consiste à reconstituer des séquences continues d'ADN chromosomique. On cherche alors à ordonner des fragments d'ADN clonés qui se chevauchent. Pour établir ces cartes physiques, les marqueurs des cartes génétiques constituent une aide indispensable.
Les cartes des gènes
Ce troisième type de cartes est particulièrement utile pour identifier les gènes impliqués dans diverses maladies génétiques. Il s'agit de cartes qui localisent le long des chromosomes un certain nombre de gènes dont la fonction n'est pas nécessairement connue, mais que l'on repère par des segments d'ADN (des "étiquettes") qui signent la présence de régions codantes, c'est-à-dire de gènes.
En octobre 1996, la revue Science a publié la dernière version de la carte des gènes humains, fruit d'une collaboration internationale de 19 équipes (dont 2 françaises). Cette carte localise plus de 16 000 gènes ou marqueurs le long des 23 paires de chromosomes humains. La base de données GENATLAS, éditée par le Professeur Jean Frézal (Hôpital Necker, Paris), quant à elle, recense environ 6 600 gènes humains fonctionnels localisés sur nos chromosomes.
On mesure le chemin parcouru quand on sait que fin 1994, à peine plus de 5 000 gènes ou marqueurs étaient positionnés, et qu'on espère parvenir à localiser plus de la moitié des quelque 100 000 gènes humains au cours des prochaines années!
Un gigantesque défi : le séquençage du génome
Le programme génome humain, qui a débuté en 1988, vise à déchiffer l'intégralité du message génétique codé dans notre ADN. Cette phase de séquençage monte actuellement en puissance : si seulement 1% du génome humain est aujourd'hui séquencé, la communauté scientifique internationale prévoit d'avoir déchiffré quelque 500 millions de caractères chimiques, soit plus de 14% de notre génome, d'ici à 3 ans.
La France, qui a occupé uneplace de leader dans les phases de cartographie génétique et physique, était jusqu'à présent absente des grands programmes de séquençage, dans lesquels les États-Unis et la Grande-Bretagne se sont investis très tôt. Elle va désormais s'y impliquer activement, grâce à la création d'un centre public de séquençage, à Evry (région parisienne).
Les enjeux médicaux liés aux progrès de la génétique
La formidable accélération des progrès de la génétique ouvre des perspectives qui suscitent de vifs et nombreux intérêts dans tous les domaines de la médecine humaine.
De ce champ foisonnant de connaissances communes, de ces procédures et de ces outils identiques, commencent déjà à naître des applications diagnostiques et des protocoles d'essais thérapeutiques qui concernent presque toute la pathologie humaine, tous les organes et des malades de tous âges. Trois grandes retombées sont attendues :
Identification de gènes responsables de maladies
Diagnostic et dépistage
Mise au point de nouveaux traitements
Identification de gènes responsables de maladies
Comprendre le mécanisme des maladies à l'échelle moléculaire, tel est l'enjeu lié à ces nouvelles connaissances.
Grâce à l'établissement de cartes génétiques toujours plus précises, ce sont plus de 1 500 gènes de maladies humaines qui, fin 1996, étaient localisés sur nos chromosomes ; en 1987, il n'étaient que 131 ; en 1991, 386 (voir tableau extrait de la base de données GENATLAS). Une véritable moisson, dont le rendement ne cesse de s'accélérer au fil des années.
Une fois qu'un gène est localisé, reste à le cloner et à déterminer sa séquence, et surtout à identifier sa fonction, c'est-à-dire comprendre le rôle de la protéine normale qu'il permet de produire. Fin 1996, environ 300 gènes de maladies humaines étaient ainsi clonés.
Reste aussi à montrer comment les erreurs d'écriture, ou mutations, qui affectent ce gène entraînent une dysfonction ou une perte de la protéine correspondante, et comment ces altérations participent au mécanisme de développement d'une maladie.
Ces études visent non seulement à identifier des gènes directement en cause dans diverses maladies génétiques, mais aussi à découvrir des gènes de prédisposition à des maladies communes : des gènes qui, en association avec d'autres facteurs de l'environnement et certains modes de vie, tendent, lorsqu'ils sont modifiés, à favoriser le développement d'une maladie.
Diagnostic et dépistage
Les applications médicales les plus immédiates liées à l'identification des gènes responsables de maladies concernent le domaine du diagnostic. En effet, il devient désormais possible, théoriquement et pratiquement, de dépister les anomalies génétiques les plus fines directement à l'origine d'une maladie héréditaire, ou qui prédisposent au développement d'une maladie acquise.
Ce dépistage peut s'appliquer à trois types de situations : le diagnostic de graves maladies héréditaires chez l'embryon (§ Diagnotic prénatal de graves maladies incurables), l'aide au diagnostic pour certaines maladies difficiles à identifier (§ Aide au diagnostic de maladies difficiles à identifier), et le dépistage des personnes à risque de développer certaines maladies (§ Dépistabe des personnes à risque de développer des maladies).
Diagnostic prénatal de graves maladies incurables
Parmi les quelque 5 000 maladies génétiques connues à ce jour, plus de 300 peuvent aujourd'hui être diagnostiquées avant la naissance, au sein des familles comportant des personnes déjà atteintes.
Un tel dépistage peut alors permettre aux parents de ne plus mettre au monde que des enfants indemnes, ou de bénéficier d'un conseil génétique avant de mettre en route une grossesse.
La pratique de ce diagnostic prénatal se trouve au coeur des grands problèmes actuels d'éthique biologique et médicale : respect de la personne humaine, intégrité de l'espèce humaine, interruption volontaire de grossesse...
Pour éviter tout risque de dérapage ou tentation d'eugénisme, le dépistage prénatal est désormais encadré en France par une loi (loi du 29 juillet 1994).
Aide au diagnostic de maladies difficiles à identifier
Par ailleurs, le recours au diagnostic génétique s'avère particulièrement utile dans le cas de certaines maladies difficiles à identifier.
Un diagnostic moléculaire permet alors de reconnaître et de classifier les anomalies moléculaires responsables des symptômes observés, parfois même de proposer de toutes nouvelles stratégies de diagnostic, comme dans le cas de certaines affections neurodégénératives (voir La génétique : Seconde partie§ Les déficits immunitaires combinés sévères).
Dépistage des personnes à risque de développer des maladies pour lesquelles il existe des stratégies préventives ou curatives pour lesquelles il existe des stratégies préventives ou curatives
Enfin, la découverte de gènes qui prédisposent à développer certaines maladies, et la possibilité actuelle de dépister certains sujets à risque, alors même qu'ils sont encore en bonne santé, constitue à la fois un progrès, lorsque la médecine est en mesure de proposer des moyens préventifs ou curatifs, et un grave problème éthique, dans le cas contraire.
De surcroît, il ne faut pas occulter le risque potentiel d'utilisation de cette information génétique à des fins non médicales, d'où l'importance d'un respect strict du secret médical.
Les problèmes particuliers posés par le diagnostic présymptomatique des maladies neurodégénératives héréditaires incurables à transmission dominante (comme la maladie de Huntingon) seront abordés en seconde partie du dossier génétique au chapitre Les déficits immunitaires combinés sévères.
Mise au point de nouveaux traitements
Parallèlement à ces nouvelles possibilités diagnostiques, l'étude des gènes responsables de maladies ouvre de nombreuses perspectives prometteuses dans le domaine thérapeutique.
Découverte de nouvelles cibles de médicaments
Tout d'abord, l'identification d'un gène (ou d'une protéine) à l'origine d'une maladie, qu'elle soit acquise ou héréditaire, offre de nouvelles pistes pour la conception de médicaments destinés à pallier les défauts ou les excès d'activité de ce gène (ou de cette protéine).
Par exemple, une fois que les biologistes ont mis en cause l'anomalie d'un "récepteur" particulier (une protéine située à la surface des cellules, destinée à recevoir un message extérieur et à le transmettre à la machinerie cellulaire interne), anomalie directement responsable d'une maladie donnée, il devient possible de concevoir et de développer, sur des bases rationnelles, des molécules, futurs médicaments capables d'agir spécifiquement sur ce récepteur, ou de suppléer la fonction défaillante de ce dernier par une autre voie.
Dans cette recherche de molécules thérapeutiques actives, les pharmacologues bénéficient aujourd'hui des progrès récents réalisés en matière de synthèse chimique et d'analyse informatique.
En effet, ils s'appuient désormais sur de nouvelles techniques de modélisation de molécules assistée par ordinateur, de conception d'analogues de synthèse de ces molécules, et sur des tests de sélection à haut débit de ces nombreux analogues.
L'ensemble de ces techniques permet de passer au "crible" de l'efficacité thérapeutique, sur une cible donnée, de très nombreuses molécules candidates en un temps record.
Stratégies de thérapie génique
Seconde perspective de retombée thérapeutique : la thérapie génique, une voie de recherche qui suscite un très grand intérêt mais qui reste encore largement à explorer.
Ici, le pari consiste à guérir ou atténuer les effets d'une maladie en réparant un gène défectueux ou en activant certains gènes utiles. La stratégie vise à introduire dans des cellules humaines (à l'exception des cellules sexuelles, ou gamètes) un gène "correcteur" ou "thérapeutique". Ce gène est destiné à pallier les défauts du gène atteint, dans le cas des maladies génétiques, ou bien à agir d'une autre façon, par exemple en aidant le système imunitaire à éliminer des cancers.
Dans tous les cas, on cherche à agir sur le génome pour faire produire à l'organisme une protéine d'intérêt thérapeutique : c'est pourquoi l'on parle aussi "d'ADN-médicament".
- Méthodes de thérapie génique
Le gène thérapeutique peut être introduit selon deux voies : dans des cellules qui ont été extraites de l'organisme pour être traitées, avant d'être réimplantées (c'est la thérapie génique ex vivo), ou bien directement dans des cellules de l'organisme, en administrant le gène au patient (c'est la thérapie génique in vivo).
Environ 80% des essais actuels sont des thérapies géniques ex vivo, qui concernent des cellules que l'on peut facilement prélever puis mettre en culture : cellules souches de la moelle osseuse, de la peau, du foie ou des muscles, ou cellules cancéreuses qui, une fois traitées ex vivo, sont irradiées avant d'être réimplantées.
Les thérapies géniques in vivo s'adressent aux atteintes bronchopulmonaires comme la mucoviscidose, aux maladies musculaires ou neurodégénératives.
Ces deux types de stratégies nécessitent le recours à des "navettes", ou vecteurs, capables de pénétrer spontanément dans les cellules cibles pour y introduire le gène thérapeutique. Le plus souvent, ces vecteurs sont des virus rendus inoffensifs : rétrovirus ou adénovirus. On les a "manipulés" de façon à leur greffer le gène correcteur ; lorsqu'ils infectent leurs cellules-cibles, ils leur fournissent du même coup ce gène correcteur.
Cependant l'efficacité et l'innocuité de ces vecteurs viraux restent encore à améliorer - Essais cliniques en cours
Une cinquantaine d'essais cliniques sont actuellement en cours en Europe, et plus de 150 aux États-Unis. Ils portent sur environ un millier de patients et concernent une trentaine de maladies : deux tiers de cancers, et diverses maladies acquises ou héréditaires comme les déficits immunitaires sévères ou la mucoviscidose.
La plupart de ces essais sont des essais de phase I, destinés à éprouver la tolérance à ces nouvelles stratégies et leur innocuité. Quelques-uns sont des essais de phase II, visant à mettre en évidence un éventuel effet thérapeutique.
La thérapie génique, aussi séduisante soit-elle dans son concept, n'en est qu'à ses premiers essais. Son efficacité clinique reste à démontrer. Trop peu de malades ont été traités dans chaque protocole, et le recul est insuffisant pour que l'on puisse analyser valablement les résultats de ces essais. Cette stratégie soulève de nombreux espoirs, mais elle est loin d'être utilisée en routine et doit encore faire l'objet, compte-tenu des difficultés à résoudre, de travaux approfondis de recherche et de développement.
